主な変異株のスパイクタンパク質における変異場所を全部可視化してみる〜新型コロナウイルス(SARS-CoV-2)〜、の巻
はじめに
なんか最近変異株の情報が多くなってきて。。。頭をすっきりさせたくてですね、
インドで流行して最近本邦でも話題になっているB.1.617系統も含めたVOC(Variants of Concern; 懸念される変異株)とVOI(Variants of Interest; 注目すべき変異株)のスパイクタンパク質上の変異についてまとめて、スパイクタンパク質のどこに変異の場所があるのかをぱっとみれるように可視化しました。(注;本記事のVOVとVOIはアメリカのCDCの分類をもとに記載しています。)
いろいろ調べてみても細かすぎたり、ざっくりすぎたり、自分が見たいちょうどいい感じの資料がなかなかないので作ってみようかしら、という。
なお、本記事では、スパイクタンパク質上の変異の場所を見ることしかしていませんので。。。
どの変異株がどこでどれだけの感染者数なの?とか、この変異によって感染性がどんだけ上がるの?とか、ワクチン効くの?とかの情報は他に詳しい情報があるかと思いますのでそちらをご参照くださいませ。
では見てみましょう。
関連記事:
変異株のN501Yってどこにあるのよ(K417NとE484Kも) 〜新型コロナウイルス(SARS-CoV-2)〜、の巻
COVID-19のワクチンの中身を詳しくみてみる、の巻
主な変異株のスパイクタンパク質における変異場所を全部可視化してみる、の巻
VOC、VOIの変異についての一覧表(2021年8月13日時点)
| Pango lineage (Nextstrain) | WHOのラベル | スパイクタンパク質のうちで アミノ酸が置換されている変異 | 最初に検出された国 |
|---|---|---|---|
| B.1.526 (20C/S:484K) | イオタ (ι) | (L5F*), T95I, D253G, (S477N*), (E484K*), D614G, (A701V*) | アメリカ |
| B.1.526.1 (20C) | – | D80G, Δ144, F157S, L452R, D614G, (T791I*), (T859N*), D950H | アメリカ |
| B.1.525 (20A/S:484K) | イータ (η) | A67V, Δ69/70, Δ144, E484K, D614G, Q677H, F888L | イギリス |
| P.2 (20J) | ゼータ (ζ) | E484K, (F565L*), D614G, V1176F | ブラジル |
| B.1.1.7 (20I/501Y.V1) | アルファ (α) | Δ69/70,Δ144,(E484K*),(S494P*), N501Y, A570D, D614G, P681H, T716I, S982A, D1118H, (K1191N*) | イギリス |
| P.1 (20J/501Y.V3) | ガンマ (γ) | L18F, T20N, P26S, D138Y, R190S, K417T, E484K, N501Y, D614G, H655Y, T1027I | 日本/ブラジル |
| B.1.351 (20H/501.V2) | ベータ (β) | D80A, D215G, Δ241/242/243, K417N, E484K, N501Y, D614G, A701V | 南アフリカ |
| B.1.427 (20C/S:452R) | イプシロン (ε) | L452R, D614G | アメリカ |
| B.1.429 (20C/S:452R) | イプシロン (ε) | S13I, W152C, L452R, D614G | アメリカ |
| B.1.617 (20A) | – | L452R, E484Q, D614G | インド |
| B.1.617.1 (20A/S:154K) | カッパ (κ) | (T95I*), G142D, E154K, L452R, E484Q, D614G, P681R, Q1071H | インド |
| B.1.617.2 (20A/S:478K) | デルタ (δ) | T19R, (G142D*), Δ156, Δ157, R158G, L452R, T478K, D614G, P681R, D950N | インド |
| B.1.617.3 (20A) | – | T19R, G142D, L452R, E484Q, D614G, P681R, D950N | インド |
| C.37 (21G) | ラムダ (λ) | G75V, T76I, R246N, Δ247-253, L452Q, F490S, D614G, T859N | ペルー |
太字のアミノ酸変異; 感染性の増加や免疫逃避に特に関連があるとされるアミノ酸残基 (L452, E484, N501, D614)の変異
(*) ; その株の中で少数派の変異
Δ ; 欠失を表す
注;最初にインドで検出されたB.1.617系統(B.1.617, B.1.617.1, B.1.617.2, B.1.617.3)はCDCではVOIですが、日本ではVOCのリストに加えられています。(2021年5月13日にCDCおよび国立感染症研究所のサイトを確認)。
注2;ラムダ株(λ)については、2021年8月13日の時点でCDCのVOC/VOIリストには記載がありませんが、注視しておこうと管理人が考えたので本記事の表に追加することにしました。
スパイクタンパク質の変異箇所をドメイン構造をみながら比較してみる
次に、上の表にある各々の変異株のスパイクタンパク質中の変異場所を、スパイクタンパク質のドメイン構造にマッピングすると下図の通りになりました。まとめてスッキリ見れるようになりました。
公衆衛生上よろしくないとされる変異(太字のアミノ酸でL452, E484, N501, D614)は、感染時にヒトの細胞上のACE2と結合する領域であるRBD付近に集中しているのがわかりますね。。。
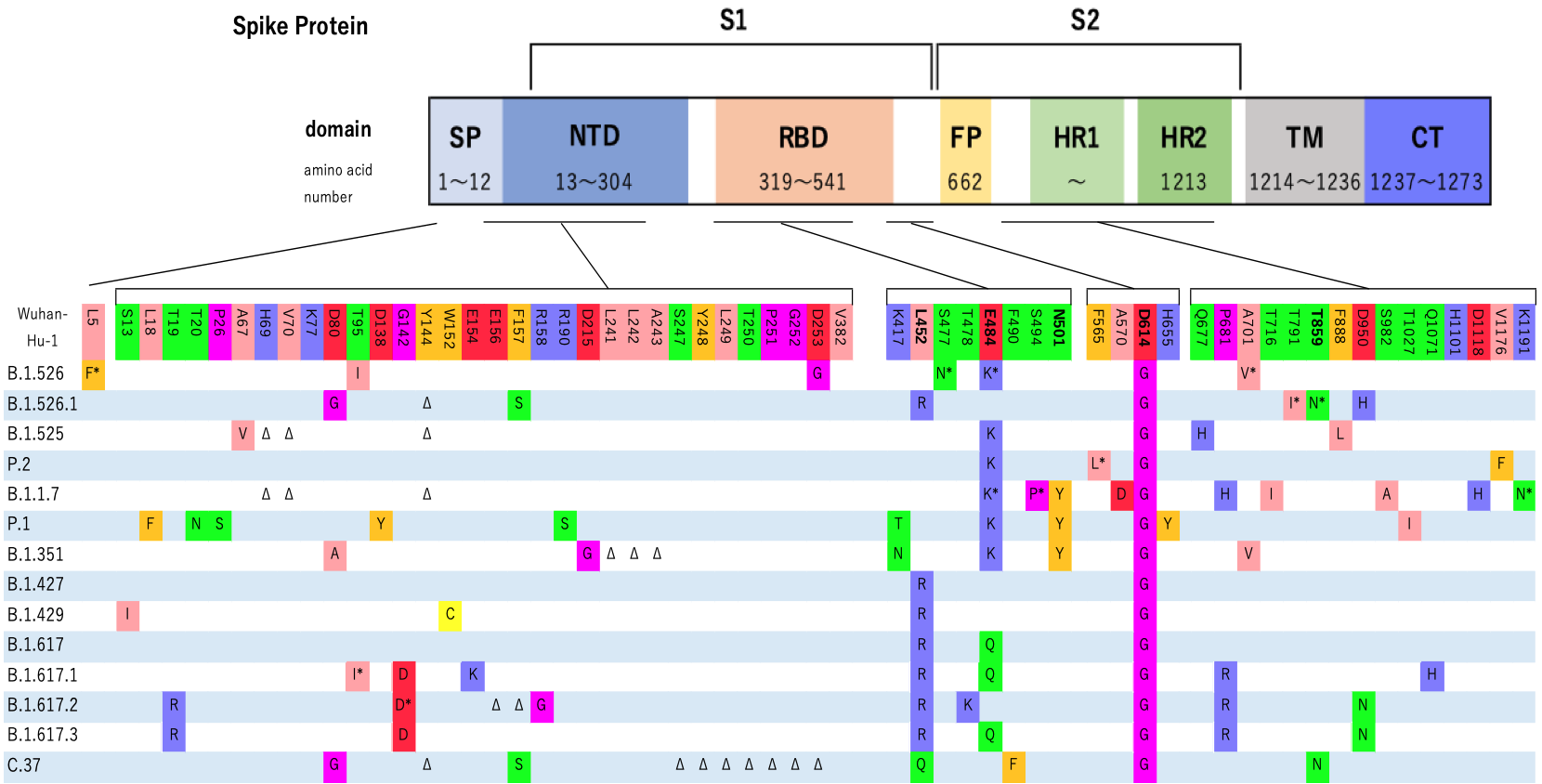
HR1; heptad repeat 1, HR2; heptad repeat 2, TM; transmembrane region, CT; cytoplasmic region.
太字; 感染性の増加や免疫逃避に特に関連があるとされるアミノ酸残基 (L452, E484, N501, D614, T859)
* ; その株の中で少数派の変異
Δ ; 欠失を表す
文献3)のFigure 1 Aを参考に作成
ちなみに上図でのアミノ酸(一文字表記)はその性質(説明)で色分けしていて、対応表は次の通りです。
| I L V A M | Aliphatic/hydrophobic |
| F W Y | Aromatic |
| K R H | Positive |
| D E | Negative |
| S T N Q | Hydrophilic |
| P G | conformationally special |
| C | Cysteine |
ふう、すっきりした。
Dash Bioでインタラクティブに見てみる
主な変異株(本記事に記載してあるやつ)のスパイクタンパク質の全アミノ酸配列を並べて比較出来るものと、三次元構造を自分で動かしながら見ることができるものをDash Bioを使って作ってみました。
全アミノ酸配列を並べて比較できるようにしたものがこちらです。
動かしながら配列を詳しく見ることができます。下側にあるのはスライダーです(スマホではスライダーが動きませんたぶん。)
下のリンクから、別windowsでもうちょっと大きく見れます。
次に、こちらはスパイクタンパク質の三次元構造に、L452, E484, N501, D614をマッピングして、三次元でどのあたりにあるのかがわかるようにしました。これも動かせます。
この構造は、スパイクタンパク質(変異前)の受容体結合ドメインにACE2(angiotensin-conberting enzyme2)の断片が結合しているところを捉えた、クライオ電子顕微鏡の単粒子解析のデータです(PDB id は7DF4です)。
スパイクタンパク質はグレー、イエロー、マゼンタで表示しています(3量体になっている)。ACE2の断片はグリーンです。このデータではACE2断片は1個だけスパイクタンパク質に結合してます。なお、簡単のため、三量体のうちでACE2と結合している分子(Chain id B)のL452, E484, N501, D614だけ色を変えています。
下のリンクから、別windowsで見れます。
おしまい。
参考文献、参考サイト
1)https://www.cdc.gov/coronavirus/2019-ncov/cases-updates/variant-surveillance/variant-info.html アクセス日2021.5.13
2)https://www.niid.go.jp/niid/ja/diseases/ka/corona-virus/2019-ncov/10220-covid19-36.html アクセス日2021.4.27
3) The influence of major S protein mutations of SARS-CoV-2 on the potential B cell epitopes
4) Cherian S, Potdar V, Jadhav S, et al. Convergent evolution of SARS-CoV-2 spike mutations, L452R, E484Q and P681R, in the second wave of COVID-19 in Maharashtra, India. doi: https://doi.org/10.1101/2021.04.22.440932
5) Pragya D. Yadav, Gajanan N. Sapkal, et al. Neutralization of variant under investigation B.1.617 with sera of BBV152 vaccinees. doi: https://doi.org/10.1101/2021.04.23.441101
6) 国立感染症研究所 新型コロナウイルス感染症(COVID-19)関連情報について
7) https://www.niid.go.jp/niid/ja/diseases/ka/corona-virus/2019-ncov/2551-lab-2/10353-covid19-44.html アクセス日2021.5.13
8) Padilla-Rojas C, et al. Genomic analysis reveals a rapid spread and predominance of Lambda (C.37) SARS-COV-2 lineage in Peru despite circulation of variants of concern, J Med Virol. 2021. PMID: 34370324
9) https://www.who.int/en/activities/tracking-SARS-CoV-2-variants/ アクセス 日2021.8.13
Dash Bio; https://dash.plotly.com/dash-bio
https://business.xserver.ne.jp/
https://www.xdomain.ne.jp/
★LOLIPOP★
.tokyo
MuuMuu Domain!

















